研究紹介
ペプチド医薬を対象としたドッキングシミュレーションプログラムの開発
医薬品開発においては大量の候補化合物から計算機を用いたスクリーニングにより化合物の生物学的な効果や作用の程度を表す「活性」が高い化合物を見出すことが行われます. 活性の指標として医薬品の標的となるタンパク質と薬剤分子の結合の強さが考えられます. 本研究では,医薬品開発におけるモダリティとして注目されているペプチド医薬に注目し,タンパク質とペプチドが結合した複合体の構造および結合の強さを評価するドッキングシミュレーションのプログラムを開発しています. 開発しているプログラムでは,タンパク質やペプチドを構成するアミノ酸残基の単位で粗視化することで高速な処理を実現し,タンパク質とペプチドの結合の強さはタンパク質構造データベースから構築した四体ポテンシャルで評価します. 高精度なスクリーニングを可能とするために,四体ポテンシャルや複合体構造の探索アルゴリズムの開発を続けています.
機械学習を用いた化合物のエネルギー・物性・生理活性予測システムの開発
医薬品開発や機能性材料の開発に向けて,分子構造と活性や物性の定量的な関係を扱う定量的構造活性相関(QSAR)や定量的構造物性相関(QSPR)が古くから研究されてきました. これらはケモインフォマティクス(化学情報学)における主要なテーマであり,近年では機械学習や深層学習の発展により活性・物性を予測する観点から注目を集めています. 本研究では,機械学習や深層学習に基づく高精度な生理活性や物性の予測システムを目指しています. さらに,分子のシュレーディンガー方程式を解いて得られるエネルギーの予測も行っています. エネルギーのよい予測モデルができれば,エネルギーの微分を用いた動力学シミュレーションやエネルギーの差分の評価により,活性や物性の高精度な予測に貢献すると期待できます.
植物工場における工場野菜の収穫量予測手法の開発
植物工場の栽培費用を削減するためには,工場野菜の生育状況に合わせて生育環境を最適化すべきです. ただし,株ごとに生育データを計測することは,手間や費用の観点で現実的ではありません. そこで本研究では,生育棚の上部に取り付けた幼苗期の生育画像を解析して将来の収穫量を予測することを目的とします. 現在は,植物の成長過程を表現する成長モデルに解析結果を適用することで予測精度の改善を試みています.
気象情報に基づく野菜市場の卸売価格予報システムの開発
施設内で生育環境を操作しながら工場野菜を栽培する植物工場は,年中通して安定した収穫が見込まれます. しかし,管理システムを終日稼働させているため,収穫量に対する収益率は乏しいです. そのため,工場野菜の販売価格は年中ほぼ一定であるため,野菜市場の卸売価格に合わせて供給量を調整すべきです. そこで本研究では,過去の気象情報および市場情報に基づいて,深層学習で将来の卸売価格を予報することを目的とします. ただし,卸売価格は週ごとに変動しやすく直接解析することは容易ではありません. 現在は,信号解析に基づいて卸売価格モデルを定義することで,複雑な変動を示す卸売価格に対しても有効な予測システムを開発しています.
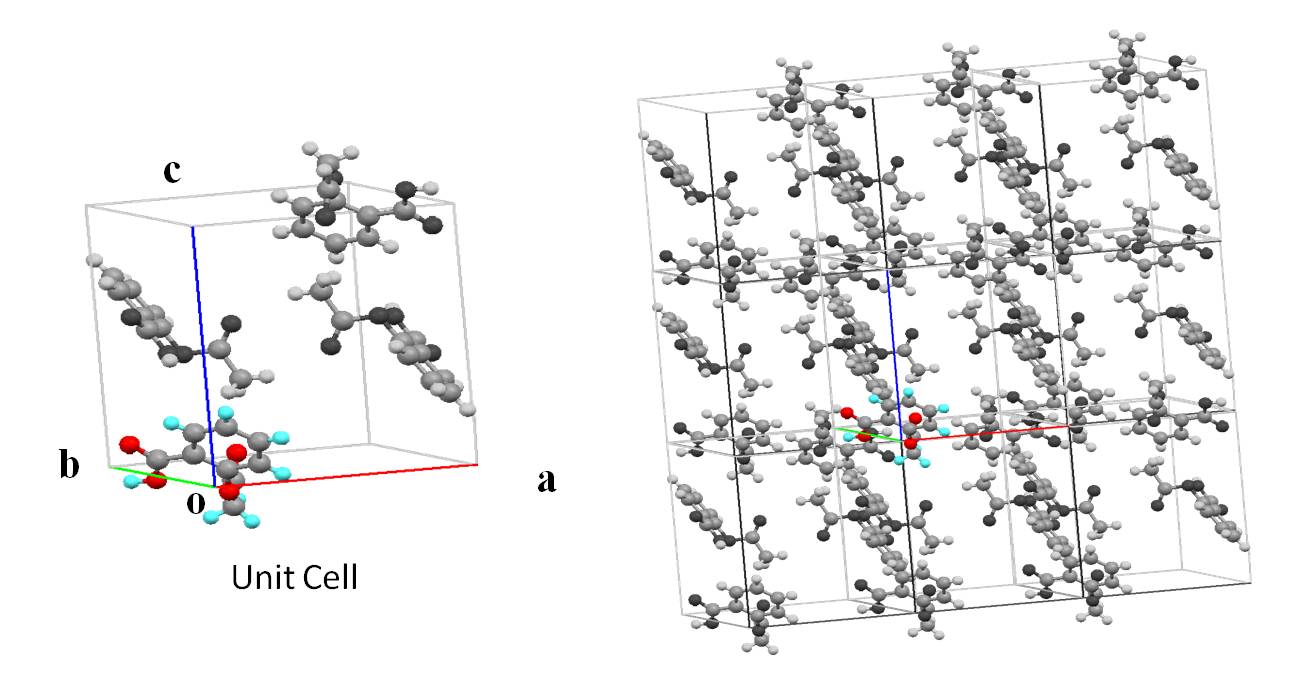
結晶多形スクリーニング技術の開発
弱い分子間相互作用からなる有機分子の結晶構造には,充填様式や配座異性体に起因する多形現象が確認されます. 結晶多形構造の違いは溶解度や電子物性などに影響するため,事前に制御できれば医薬製剤技術や新規素材開発への貢献が期待されます. 結晶構造を計算により予測するコンテスト(Crystal Structure Prediction Blind Tests)は数年ごとに開催されに行われ国際的にも注目されています. 本研究では,分子力場による球状微細結晶モデルを使った結晶構造を最適化するCONFLEXを開発しています. このプログラムにより種々の有機分子に対して結晶多形を網羅的に探索でき,条件にあてはまる結晶を選別するスクリーニングが可能となります.
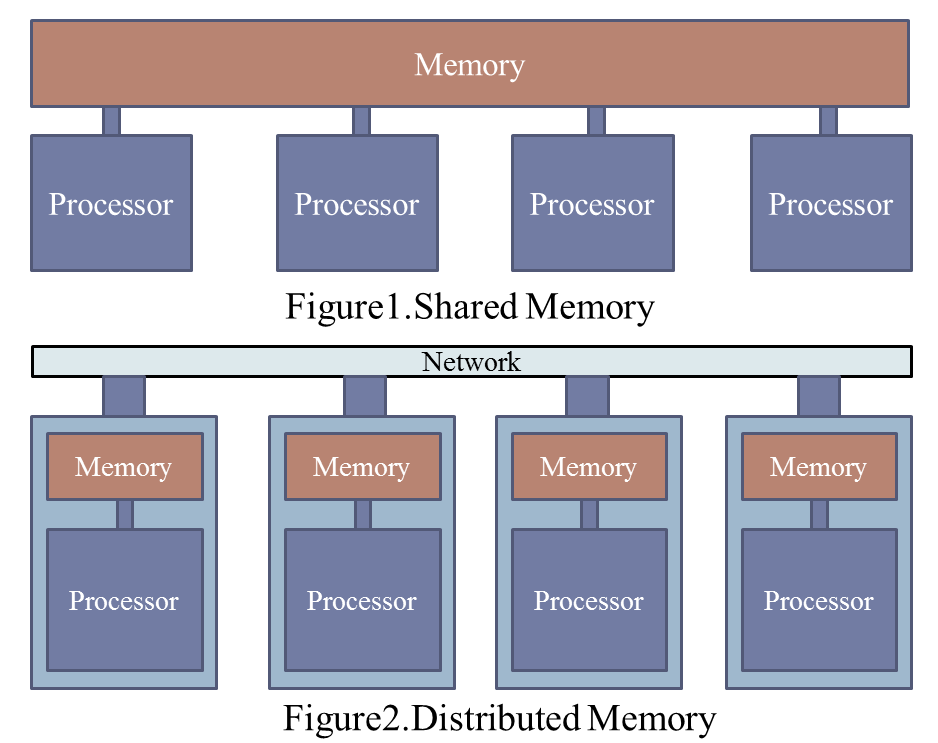
並列分散処理技術を用いた結晶計算の高速化
結晶計算はナノ科学や創薬,機能性材料開発など多方面での活用が期待されますが,多数の分子を含む分子集合体を解析するためには多大な計算量を要します. 本研究では,分子の配座探索や結晶構造探索を行うプログラムCONFLEXに分散メモリ型並列処理技術であるMPIを適用し,高速な計算を実現しました. さらに,共有メモリ型並列処理技術OpenMPとのハイブリッド型並列処理へ展開して結晶多形の探索を高速化しました.